Medical Data Lake
Nutzung des medizinischen Datenschatzes zur Erhöhung der Behandlungsqualität
Ist es ein gutes Gefühl, einen Schatz zu besitzen, den man nicht heben kann? Diese Frage stellt sich vielen Krankenhäusern, die einen medizinischen Datenschatz besitzen und diesen verwalten, ohne ihn aber effektiv nutzen zu können. Ralph Szymanowsky geht in diesem Artikel der Frage nach, wie das umfangreiche Wissen, das über viele Jahre in verschiedensten Datensystemen in Krankenhäusern gesammelt und gespeichert wurde, in einem Medical Data Lake zu einem organisierten und flexiblen Wissensreservoir wird, das zur Verbesserung von Behandlungen genützt werden kann.
„Daten sind das neue Öl“ ist ein typisches Buzzword, aber durchaus mit einem wahren Kern. Öllagerstätten können schwer zugänglich und der Bodenschatz somit kaum verwertbar sein. Die Förderung des Rohöls bedeutet nicht automatisch ein hochwertiges Produkt, sondern erfordert eine aufwändige Verarbeitung, ganz abgesehen von unterschiedlichen Qualitäten des Rohöls – und auch der Daten.
Über Jahre wurde umfangreiches Wissen über verschiedenste Krankheitsbilder gesammelt. Die Dokumentation zu einzelnen Patienten wurde akribisch angelegt und enorme Summen für die Diagnostik ausgegeben, nichtsdestotrotz sind die daraus resultierenden Erkenntnisse meist auf den Erfahrungsgewinn einzelner Behandler und die Genesung der Einzelpatienten begrenzt. Erschwerend kommt hinzu, dass die Dokumentation zu einzelnen Patienten weniger die übergreifende Auswertung der Daten, als vielmehr die schnelle Verfügbarkeit im Patientenkontext zum Ziel hatte. Der Aufbau des Datenmodells ähnelt dem einer Bibliothek. Man findet die Werke einzelner Autoren und Genres schnell, allerdings wird es kompliziert, wenn sich die Frage auf Inhalte von Werken bezieht, wie z.B. „alle Romane, in denen von Epidemien die Rede ist“. Hier ist man weitgehend auf die Erfahrung und das Erinnerungsvermögen des Bibliothekars angewiesen, entsprechende Suchalgorithmen gibt es nicht – was mit Blick auf die Aufgabe von Bibliotheken auch nachvollziehbar ist.
Mit dem Medical Data Lake wird aus der starren Daten-Bibliothek ein neu organisiertes und flexibles Medizinisches Wissensreservoir.
Unsere Vision ist eine ganzheitliche Betrachtung der Daten in einem Medical Data Lake inklusive Labor, Diagnostik, Befunddokumentation, Medikamente, Monitoring, EKG etc., um nicht nur Kosten, Prozesse und Ressourceneinsatz zu optimieren, sondern auch für die klinische Qualität Erkenntnisse ziehen zu können. Die Auswertung aller im Krankenhaus verfügbaren Daten sollte Erkenntnisse zur Verbesserung der Behandlung, Evaluierung von Behandlungsrichtlinien und Aussagen zur Behandlungsqualität möglich machen.
Als technologische Grundlage wurde dabei bewusst auf FHIR* gesetzt. Diese Technologie unterstützt den Datenaustausch zwischen Softwaresystemen im Gesundheitswesen über sogenannte „Ressourcen“.
Ziel dabei ist die Schaffung einer medizinischen Datenbasis zur Charakterisierung von Patientenpopulationen anhand verschiedener Parameter wie
- demografische Informationen
- Krankheitsverläufe
- Labor
- Medikation
- Morbidität
- Behandlungen und Behandlungsabfolge usw.
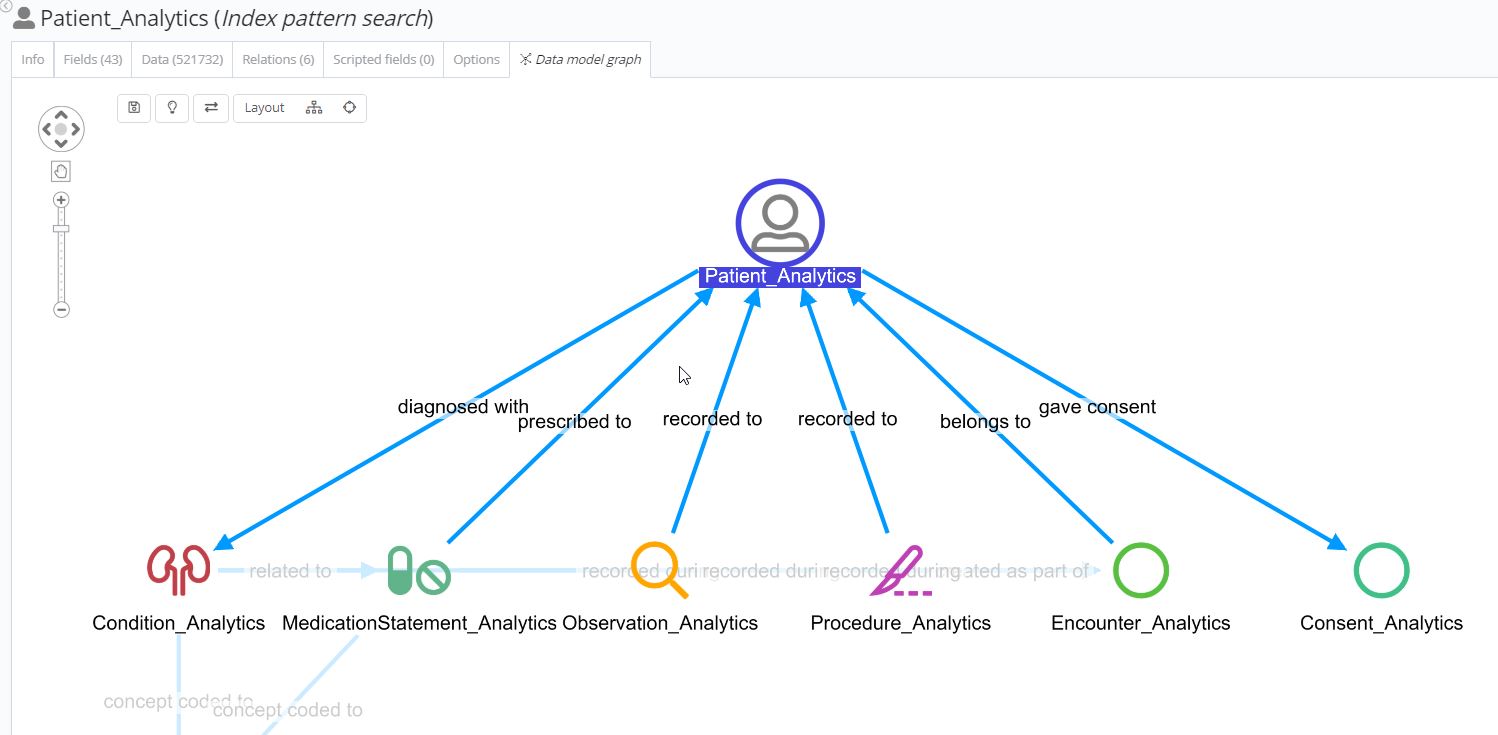
Die an OMOP** angelehnte herstellerunabhängige Organisation der Daten richtet sich dabei an der medizinischen Analyse aus und ist personenzentriert aufgebaut. Alle klinischen Ereignistabellen sind mit der Tabelle / Ressource Person verknüpft.
Meilensteine auf dem Weg zum Medical Data Lake
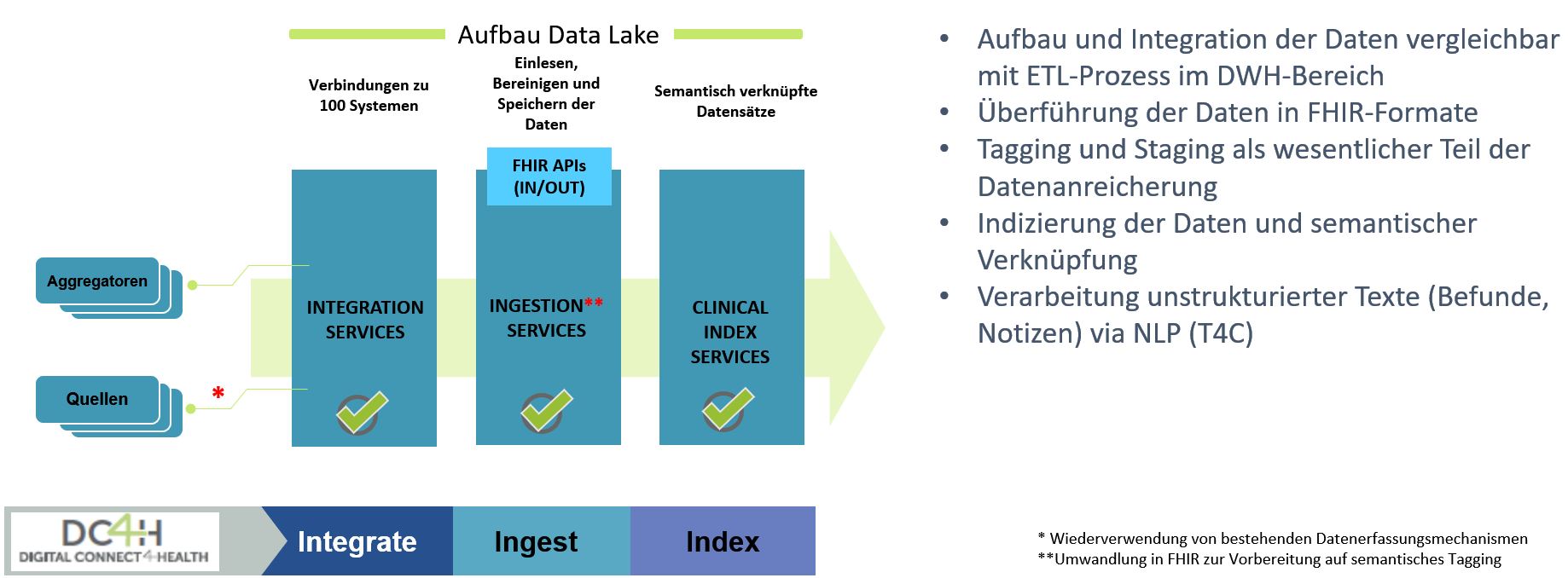
1. Integration
Entsprechend der aktuell abgebildeten Ressourcen (Person, Observation, Condition, Drug, Measurement) werden die entsprechenden Rohdaten per SQL (vergleichbar dem ETL-Prozess für BI-DWH) aus den KIS-Systemen extrahiert. Die Dedalus-Systeme (ORBIS, DXCare, Lorenzo und Galileo) sind bereits integriert. Die umfangreiche Erfahrung von TIP HCe in der Anbindung entsprechender Systeme ermöglicht den zügigen weiteren Ausbau und die Integration weiterer KIS/KAS/EMR.
2. Ingestion
Dies beinhaltet die Überleitung der Daten in FHR-Formate und in das an OMOP angelehnte Datenmodell. Dieser Schritt ist zum einen Voraussetzung für die flexible Analyse der Daten und zum anderen eine Möglichkeit, zukünftig direkt Daten aus den verschiedenen Systemen via FHIR übernehmen zu können bzw. Daten anderen Systemen via FHIR zur Verfügung zu stellen.
3. Index
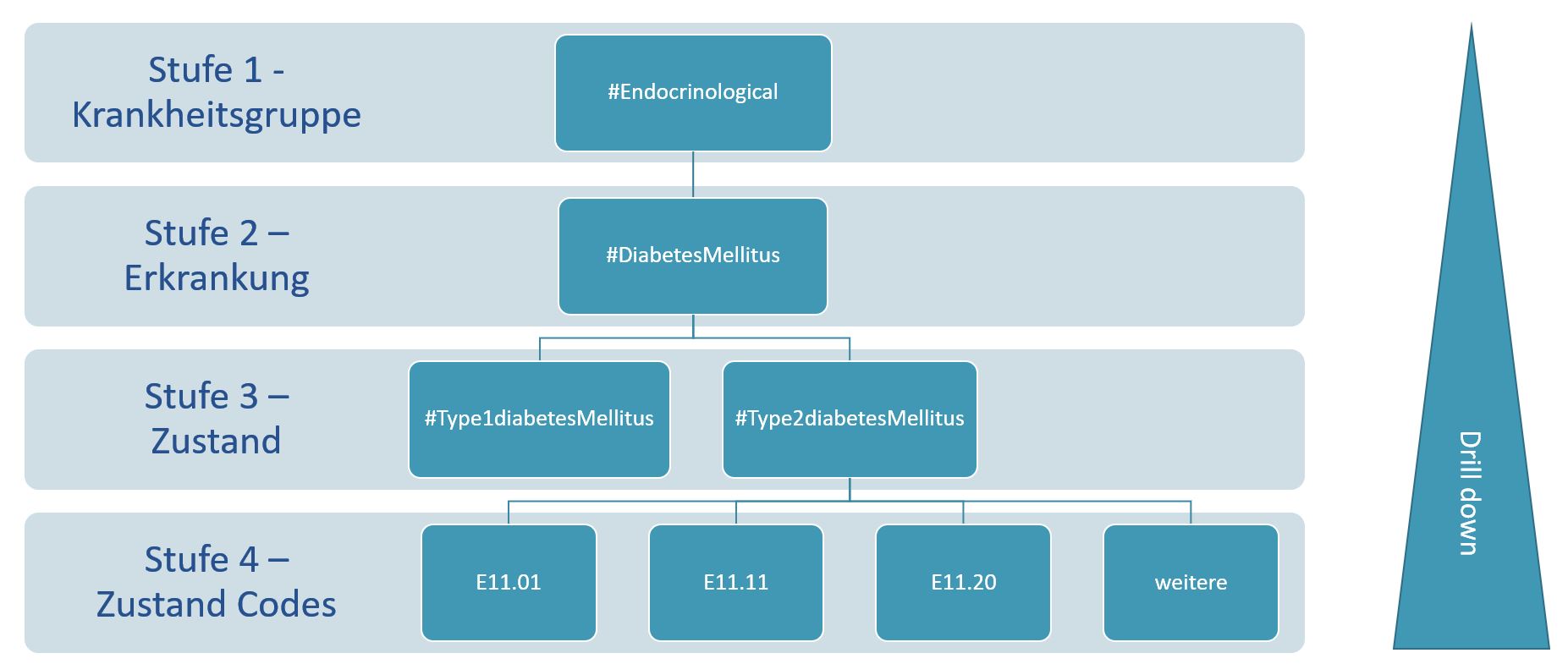
Das Tagging und Staging der Daten ist Teil der Datenanreicherung um analyserelevante Kommentierungen der übernommenen Rohdaten. Es reicht nicht aus, in Befunden nach Schlagworten oder prägnanten Formulierungen zu suchen, da deren Kontext sich aus dem Gesamttext ergibt und für die korrekte Suche nicht ausreichend ist. Beispiel: Die Suche nach ‚Diabetes‘ in der Dokumentation zum Patienten kann zu Suchergebnissen von Texten mit Inhalten wie ‚Keine Diabetes‘ oder ‚Verdacht auf Diabetes‘ sowie ‚Diabetes Typ 1‘ führen und ist somit alles andere als zielgenau. Das Tagging reichert die Daten zum Patienten mit hierarchisch strukturierten Tags automatisch an. Dieses Verfahren ist jedoch nicht auf Conditions beschränkt, sondern entsprechende Methoden werden für Observation (z.B. Laborwerte – pathologisch etc.) und Drug (Medikation – Wirkstoffe etc.) sowie Procedures angewendet.
Analyse-Möglichkeiten und Ausbau
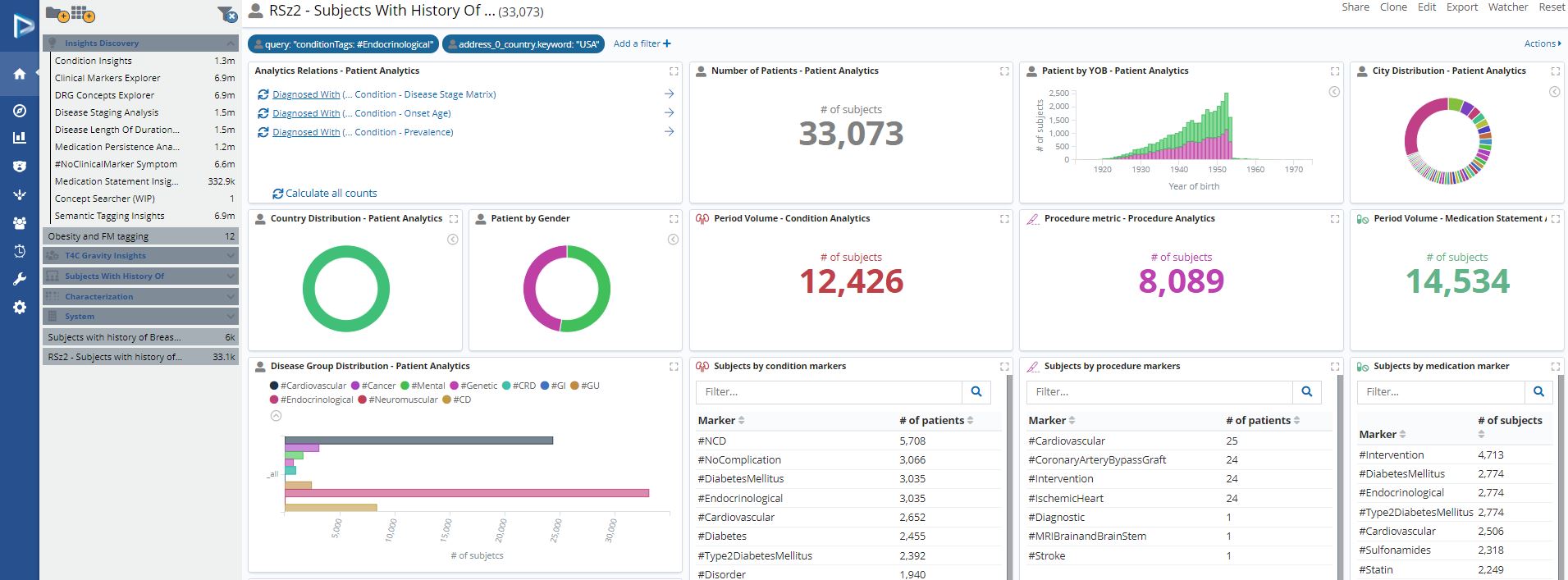
Die Analyse der Daten wird dem Anwender über Dashboards ermöglicht, dabei besteht an verschiedenen Stellen die Möglichkeit des Exports von Detaildaten. Die einfache Definition und Kombination von Filtern wird über die GUI unterstützt, darüber hinaus besteht die Möglichkeit, mittels Expertenfunktion komplexere Bedingungen zu formulieren.
Die Dashboards können intern bereitgestellt und mit wenigen Schritten auf spezielle Krankheitsbilder adaptiert werden, was die breite Nutzung auch durch weniger IT-versierte Kollegen möglich macht.
Geeignet ist diese Form der Datenanalyse insbesondere für leitende und forschende Ärzte, Apotheker, Hygieniker, Medizincontroller und die Mitarbeiter der Qualitätssicherung.
Der „Datenschatz“, welcher mit diesen Schritten gehoben wurde, ist für die Beantwortung folgender interner Fragestellungen hilfreich:
Erhalten Patienten mit Nierenschädigung auch immer die passende Medikation?
Dauer von Antibiosen bei verschiedenen Krankheitsbildern
Einsatz von Antibiotika nach Krankheitsbild
Auch zur Weiterentwicklung des medizinischen Spektrums sind die Daten hilfreich, wenngleich „nur“ die Daten der eigenen Institution optimal strukturiert zur Verfügung stehen. Das bedeutet letztlich, die Forschung zu seltenen Erkrankungen o. Ä. wird in den meisten Einrichtungen damit an Grenzen stoßen.
Research und T4C (Life Science)
Die medizinische Forschung zum Beispiel zu seltenen Erkrankungen bzw. die Bildung von Patientenkohorten zu umfassenden klinischen Studien bedingt in vielen Fällen eine größere Zahl von Patienten inklusive umfassender Dokumentation und je nach Fragestellung weitere Fachrichtungen.
Wenn man diesem Weg konsequent folgt, kann ein Krankenhaus Teil eines größeren gemeinsamen Data Lake oder Research Hub werden und gleichzeitig die Hoheit über die eigenen Daten behalten.
Dieser Bereich würde insbesondere wegen der Behandlung von Fragen des Datenschutzes und der Ethik den Rahmen sprengen und wird daher hier nicht weiter ausgeführt. Falls Sie Fragen zu diesen Möglichkeiten haben, melden Sie sich bitte mit dem Verweis auf das Thema Life Science.
Artikel vom 24. November 2021